北京擎科生物科技股份有限公司品牌商
7 年
手机商铺
- NaN
- 0.7000000000000002
- 0.7000000000000002
- 1.7000000000000002
- 0.7000000000000002
北京擎科生物科技股份有限公司
入驻年限:7 年
- 联系人:
张楠
- 所在地区:
北京 大兴区
- 业务范围:
实验室仪器 / 设备、试剂、技术服务、细胞库 / 细胞培养、抗体、ELISA 试剂盒、耗材
- 经营模式:
生产厂商 科研机构
公司新闻/正文
基于FACS的单B细胞筛选平台加速抗体发现
615 人阅读发布时间:2026-01-20 14:32
自1986年单克隆抗体Orthoclone OKT3(muromonab-CD3)获批以来,尤其自1994年重组抗体Abciximab(Reopro®)问世后,单克隆抗体(mAbs)已成为现代治疗的重要支柱。截至2024年,FDA共批准159种抗体类生物制剂,其中治疗性单抗超过100种,涵盖抗体药物偶联物、双特异性抗体及Fc融合蛋白等多种形式[1]。
一、单B细胞筛选的崛起
抗体发现主要依赖多种经典筛选技术,包括单B细胞筛选、杂交瘤、噬菌体展示及转基因小鼠等方法。在全球抗体发现市场中,单B细胞筛选技术于2024年约占10%的市场份额[2],预计在2025至2033年间将以约14.7%的复合年均增长率(CAGR)持续增长[3]。其中,单B细胞筛选近年来发展迅速,表现尤为突出。一项研究表明,经抗原特异性富集的单B细胞中,功能性抗体比例可达72%,显著高于噬菌体展示法的39%[4]。该技术能够直接从免疫动物或人源供体中分离抗原特异性B细胞,并保留天然的重链(VH)与轻链(VL)配对,从而有效提升抗体的功能完整性与亲和力。
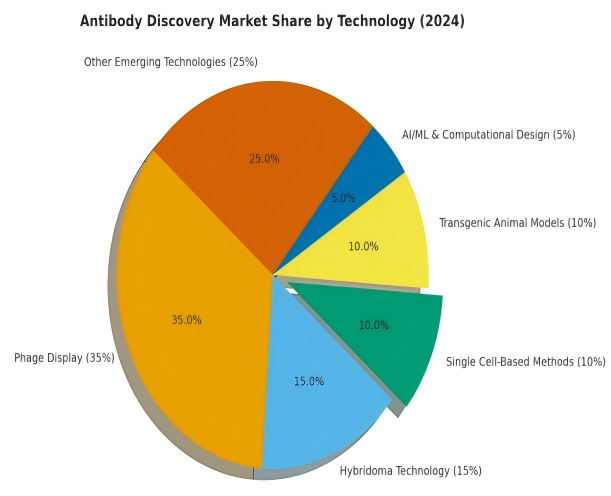
图1 2024年抗体发现市场各技术占比[2]
二、单B细胞筛选的核心技术
在单B细胞抗体发现领域,荧光激活细胞分选(FACS)依然是核心技术,因其在速度、精确度和多参数分析方面具有无可比拟的优势。通过多色荧光标记抗原和细胞表面标记物,FACS能够以每秒高达一万细胞的速度快速分离单个抗原特异性B细胞,非常适合大规模、高通量筛选。其在高特异性捕获稀有细胞群体方面的能力,加上成熟的实验方案和广泛的可用性,使其在不同研究环境中可靠且适应性强。虽然基于微流控的系统(包括微滴和微孔平台)在进行分泌抗体功能分析方面表现出色,但通常伴随更高的器件复杂度、成本及下游回收效率挑战。相比之下,FACS流程简洁、体系成熟,尤其适用于记忆B细胞的抗原特异性筛选,可直接获取用于基因扩增的单细胞样本[5]。
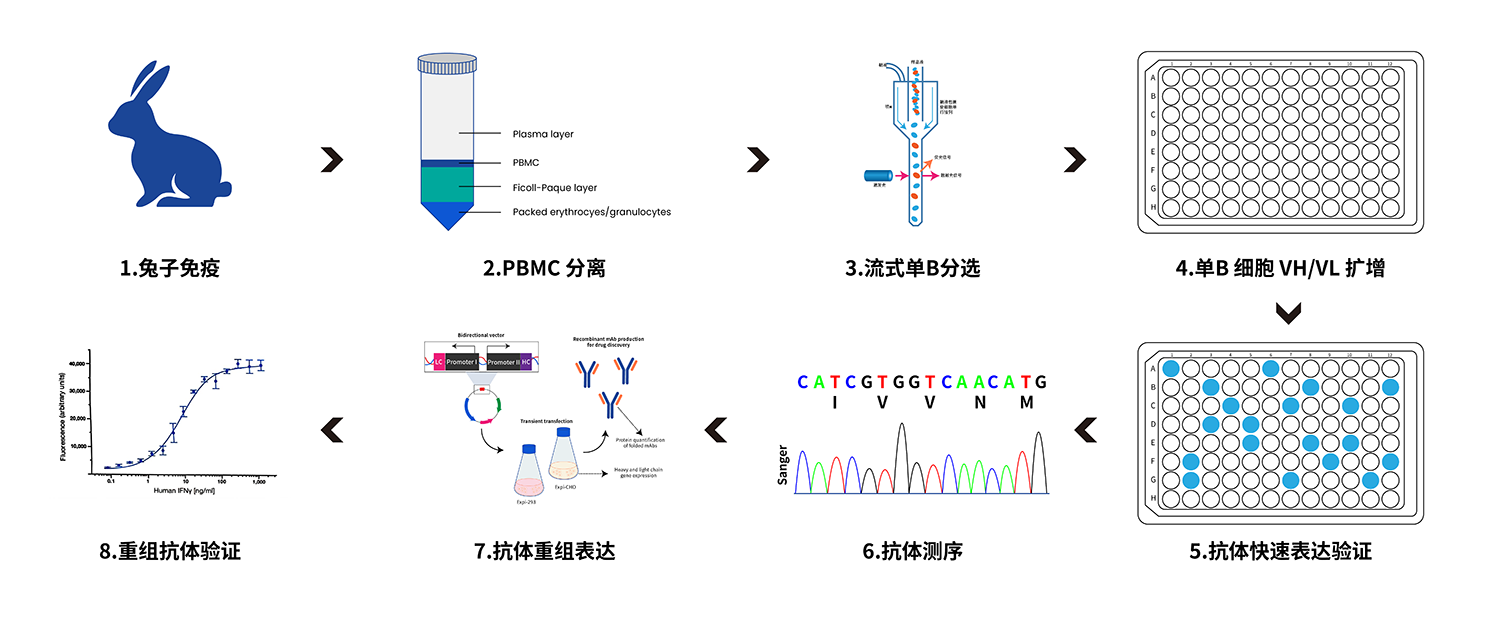
图2 基于FACS的单B细胞抗体筛选流程图
三、单B细胞筛选流程
单B细胞筛选流程始于对模型动物(如:兔子)进行抗原免疫,以激发抗原特异性B细胞反应。随后分离外周血单个核细胞(PBMCs),并通过FACS精确筛选抗原特异性B细胞。随后对这些B细胞的VH和VL基因进行RT-PCR扩增,并克隆入表达载体进行瞬时抗体表达。表达的抗体经测序以确认其序列及多样性,然后在哺乳动物细胞系(如HEK293或CHO细胞)中进行大规模表达。最后,通过ELISA、表面等离子共振(SPR)或细胞实验等方法对抗体进行功能验证,包括结合力、特异性及活性,从而实现高亲和力、抗原特异性的单克隆抗体快速发现与开发。
服务优势
更快:仅需8–10周,比传统杂交瘤法缩短约50%。
更准:天然VH/VL配对,保证抗体的结构和功能真实性。
更强:筛选得到的抗体亲和力高达10-⁸–10-¹² M。
更稳:鼠源与兔源系统兼容,满足药企、诊断公司及科研客户多样需求。
服务详情
|
服务名称 |
服务编号 |
周期 |
交付 |
|
单 B 细胞抗体筛选服务 - 鼠单抗 |
Tsingke-B01
|
8–10 周
|
阶段 I 提供每只动物血清 5 μL,检测效价;若 ≥1:100 K,则选取最佳个体进入阶段 II 阶段 II (1) 10株阳性上清 (2) 1株抗体基因序列 (3) 1–3 mg 纯化单抗及对应表达质粒(ELISA 效价 ≥1:100 K)
|
|
单 B 细胞抗体筛选服务 - 兔单抗 |
Tsingke-B02 |
擎科加速您的抗体发现之旅
擎科抗体一站式服务,为您提供从免疫原到抗体开发的全流程解决方案。
擎科生物单B细胞抗体筛选平台,已准备就绪,助您快速解锁高亲和力抗体的巨大潜力。
立即联系我们:protein@tsingke.com.cn,了解如何利用我们的平台优势,显著缩短您的候选分子发现周期。
[1] A correction has been published: Antibody Therapeutics, Volume 8, Issue 3, July 2025, Page 238
[2] Antibody Discovery Market Future Shaped by AI and Partnerships
[3] Single B Cell Antibody Discovery Market
[4] Mahendra, A., Haque, A., Prabakaran, P. et al. Honing-in antigen-specific cells during antibody discovery: a user-friendly process to mine a deeper repertoire. Commun Biol 5, 1157 (2022).
[5] 10.1186/s13036-025-00513-z







